O aprendizado de máquina – sub-área da Inteligência Artificial (IA) – é uma tecnologia já presente no nosso dia a dia. Ela está nos filmes e séries, nas detecções de fraudes bancárias e até mesmo no diagnóstico de doenças como o câncer.
Essa tecnologia combina a ciência da computação com a estatística e permite que computadores “aprendam” com dados, identifiquem padrões relevantes e não triviais e tomem decisões sem necessidade de explicitamente serem programados para isso.
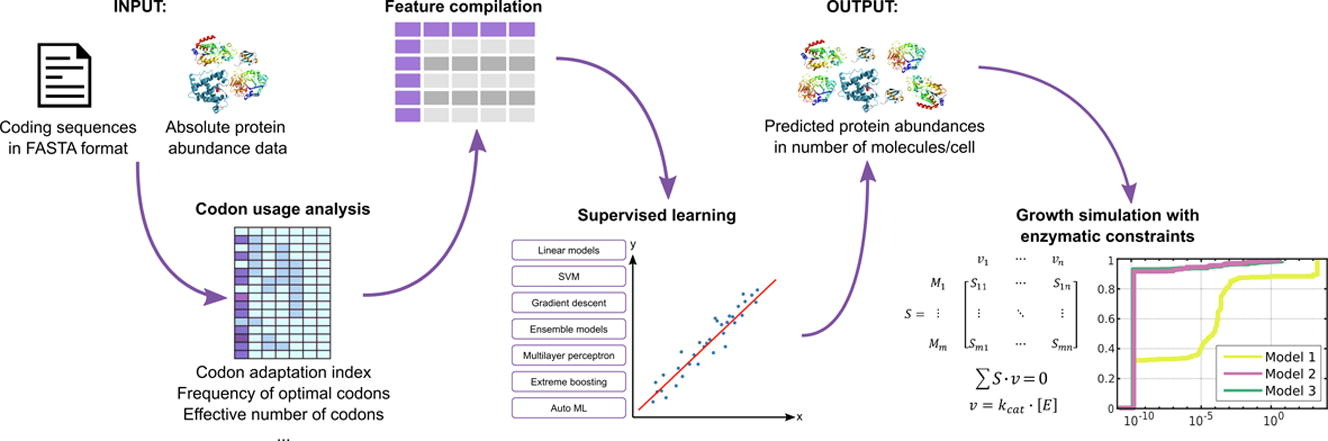
O trabalho intitulado Protein Abundance Prediction Through Machine Learning Methods utilizou estratégias de aprendizado de máquina para adquirir conhecimento sobre a quantidade de proteínas em uma célula. Isso é importante para diferentes áreas, por exemplo a engenharia de proteínas recombinantes, que pode utilizar essa informação para guiar processos utilizados pela indústria farmacêutica para produção de diversas proteínas terapêuticas como a insulina, interferons e anticorpos monoclonais, utilizados para tratar doenças como diabetes, câncer e HIV. Também é importante para a engenharia metabólica, empregada para gerar novas linhagens de microrganismos com maior capacidade de produção de biocombustíveis e outros bioprodutos de interesse industrial como aminoácidos, ácidos orgânicos, vitaminas, poliálcoois e pigmentos.
Os responsáveis pelo trabalho foram os pesquisadores Maurício Ferreira, Rafaela Ventorim, Eduardo Almeida, e Wendel Silveira, do Programa de Pós-graduação em Microbiologia Agrícola, e Sabrina Silveira do Programa de Pós-graduação em Ciência da Computação, todos da UFV. A pesquisa foi publicada no periódico Journal of Molecular Biology e nasceu a partir da dissertação de mestrado do autor Maurício Ferreira, orientada pelo Prof. Wendel Silveira.
No trabalho conduzido pelos pesquisadores da UFV, estratégias de aprendizado de máquina foram aplicadas para predizer a quantidade de proteínas que existem dentro de uma célula utilizando informações contidas nas próprias sequências de DNA de um organismo, chamadas de códons. Os códons são conjuntos de três nucleotídeos que codificam os aminoácidos que farão parte de uma proteína, e são usados pela célula de forma diferente dependendo da quantidade de proteína que um determinado gene é responsável por produzir. Proteínas que existem em alta quantidade possuem um padrão de códons diferente de proteínas que existem em baixa quantidade, hipótese que deu base ao trabalho. Além disso, para demonstrar que as predições de quantidade de proteínas realizadas por meio de estratégias de aprendizado de máquina possuíam valor biológico, simulações computacionais do metabolismo de leveduras foram realizadas incorporando os dados preditos.
O periódico Journal of Molecular Biology, onde foi publicada a pesquisa, possui diversos artigos na área de biologia molecular e genética, além de publicações figuras históricas como Jacques Monod, que descreveu o operon lac e a diauxia em bactérias, Edwin Southern, inventor da técnica de Southern Blot, Temple Smith e Michael Waterman, que criaram o alinhamento de sequências de DNA, e Stephen Altschul, co-criador do BLAST, a ferramenta de bioinformática mais utilizada por pesquisadores.





Deixar um comentário